

群落多样性

基因功能挖掘

快速

全面


小片段文库

PE150测序

《肝硬化中人体肠道菌群的变化》
肝硬化在世界范围内普遍存在,许多慢性肝病最后会发展为肝硬化。本文通过对98例肝硬化患者和83例健康对照者的比较,对肝硬化患者的肠道微生物群进行了描述,并建立了参考基因集。
研究方法:
取材:收集了中国汉族123例肝硬化患者和114个健康人粪便样本,其中实验阶段98个肝硬化患者及83 个健康人的粪便样本;验证阶段收集了 31个健康人和25个肝硬化患者的粪便样本。
测序:Illumina HiSeq2000测序平台(PE100),平均每个样本4.75GB数据量。
分析:数据质控、序列拼接组装、基因集构建、群落结构组成差异分析、基因丰度分析、MGS分析、组间功能和通路差异分析、疾病诊断模型建立。
研究结果:
◎建立肝硬化患者肠道菌群基因集,分析得到肝硬化患者与健康人的菌落结构存在显著差异,参与的信号
通路也有差异。
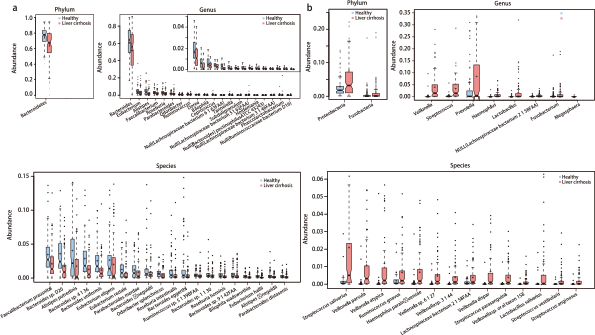
图2. 肝硬化病人和健康人在不同分类水平上的物种丰度差异。
相对于健康人,在门、属、种水平上肝硬化病人丰度减少的物种(a)和丰度增加的物种(b)
◎ 通过聚类分析,寻找与肝硬化密切相关的细菌,共找到66个物种(MGS),其中38个细菌在健康人中富集,
28个细菌在肝硬化病人中富集。

图3. 分别在肝硬化病人和健康人中富集的物种
◎基于mRMR方法寻找到15个高特异性和灵敏度较高的微生物基因标志物,建立了识别肝硬化病人的诊断模型
(PDI),在验证阶段,经分析能够利用此模型有效鉴定肝硬化患者。
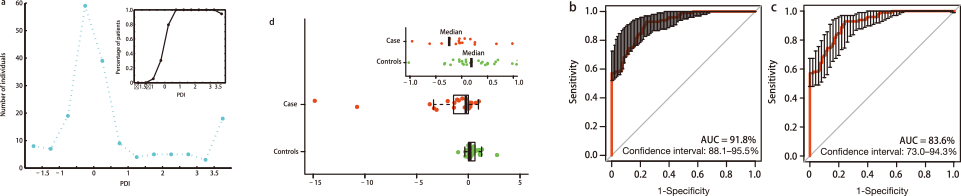
图4. 基于标记基因物建立疾病诊断模型,该模型能有效辨别肝硬化病人和健康人
肝硬化参考基因集包含269万个基因,36.1%是之前未公布的。通过与2型糖尿病和炎症性肠疾病基因集的比较,揭示了针对肝硬化病人基因和功能水平的生物标记物。找到一种只有15个基因构成生物标记物作为一个高精确的区分病人的指数。因此,微生物靶向生物标志物可能是一个用于诊断不同疾病的强有力工具。
参考文献:
Nan Q, Fengling Y, Ang L, et al. Alterations of the human gut microbiome in liver cirrhosis. Nature.2014.